一方、副甲状腺ホルモン(PTH)の添加により、MBD4がリン酸化され5mCpG領域にAPサイトが形成される。
その後塩基除去修復機構により脱メチル化される。その結果、CYPB27B1の発現が回復される。
−東京大学、オックスフォード大学研究チームが米国科学アカデミー紀要に発表−
このたび、東京大学先端科学技術研究センター(以下、東大先端研)の和田洋一郎特任研究員、大田佳宏特任研究員、井原茂男特任教授、児玉龍彦教授(システム生物医学分野)らは英オックスフォード大学と連携して、「ヒトの染色体にポリメレースの複合体が結合してRNAが作られてゆく様子をとらえる」ことに成功しました。本成果は、日本時間10月13日(火)午前6時に米国科学アカデミー紀要の電子版に発表されました。
Journal website (OPEN ACCESS ARTICLE)
PubMed
概要:
遺伝子のDNAの配列を鋳型にRNAポリメレースという酵素によってRNAが作られ、RNAの配列をもとにタンパク質が作られることは、生命の基本原理と考えられています。しかし、ヒトの細胞核内のDNAは、タンパク質の情報をもつ部分が飛び飛びに存在し、しかもDNAがヒストンというタンパク質にまきついて染色体を作るため、DNAからRNAが作られるところを実際に観察するのはこれまで困難でした。ところが最近、ゲノムの解読からヒトの染色体のもつDNAの配列が明らかになり、RNAが作られて行く全体像を解読することが可能となってきています。
東大先端研の和田洋一郎特任研究員、大田佳宏特任研究員、井原茂男特任教授、児玉龍彦教授は英国のオックスフォード大学と連携して、ヒトの血管の細胞が炎症の刺激をうけた後、7.5分おきにRNAが作られて行く様子を染色体上で観察、ポリメレースが3100塩基(約1ミクロン)/分で動き、未熟なRNAが切断されると同時に次々と作られていく過程を観測することに成功しました。
通常DNAはコヒーシンおよびCTCFというタンパク質がともに作用し、束ねられ、ポリメレースは活性化される前からこのコヒーシンで区切られた染色体上の特定の狭い領域に集まっていますが、活性化されるとそこから動きだし、さらに先のコヒーシン(およびCTCF)の部分でスピードダウンしながら進んでいくことを見いだしました。従来ポリメラーゼは確率的に動いたり止まったりすると考えられていましたが、今回コヒーシンがこの動きを制御していることを明らかにしました。
[具体的な成果]
遺伝子の発現の時間依存性を精密に測定し、大量データの詳細な解析から導いた結果に対し、さらに実験で確認するという実験と計算のタイトな連携によって発見につなげました。
1)ポリメレースが作るRNAを短い時間間隔で計測
(特色)背景には空間分解能の高いカスタムのタイリングアレイ、細胞刺激の実験系が整備、それによる高精度精密解析(実験と情報解析)を行った。
2)実体としてのポリメレースの解析から新しい発見
3)意義
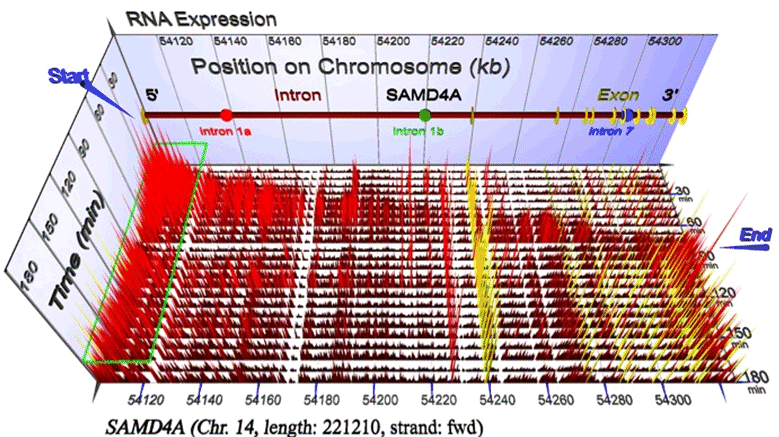
図1 RNAの転写が遺伝子上を波のようにSAMD4A遺伝子を伝搬していく様子 RNAプローブを遺伝子にしきつめたタイリングアレイで計測し、転写の全体像を得た。
遺伝子の位置を横軸に、手前の軸に時間、上向き軸にRNAの発現の強さを表示。遺伝子はイントロン部分とエクソンの部分からなり、イントロン部分のRNAを赤で、イントロン部分を黄色で示す。イントロン部分のRNAは時間が経つと消滅し、エクソン部分のRNAは時間とともに蓄積されているようにみえる。 刺激直後(start)から(end)まで、RNAの転写が遺伝子上を波のように遺伝子を伝搬していき、安定に転写をする状態になっていく。
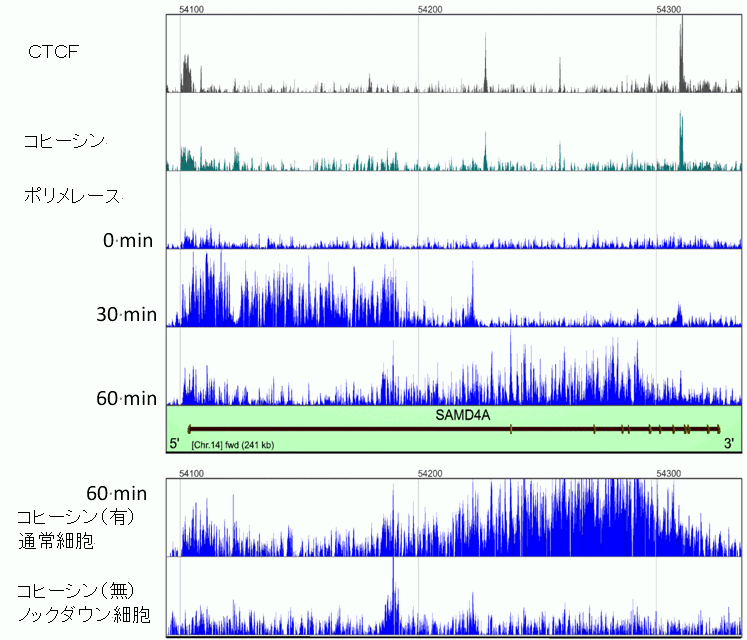 図2 コヒーシンの有無で転写をつかさどるポリメレースの運動が変わる様子
遺伝子上でポリメレースの濃度を測定できる抗体を用い、網羅的にチップーチップ法で検出し、ポリメレースの運動を追跡した。
図2 コヒーシンの有無で転写をつかさどるポリメレースの運動が変わる様子
遺伝子上でポリメレースの濃度を測定できる抗体を用い、網羅的にチップーチップ法で検出し、ポリメレースの運動を追跡した。上の図で、横軸は遺伝子、縦軸はそれぞれの分子の濃度を表す。 刺激後、ポリメレースは遺伝子の位置とその0分、30分、60分と移動していく。 コヒーシンが有るとき、ポリメレースはコヒーシンのあるところで停留し、濃度差が大きくなる。一方、コヒーシンが無いとき、ポリメレースの濃度差は遺伝子全体で平坦になっていくことがみてとれる。
今回の成果によって、ヒトの複雑な染色体上でRNAが作られる様子が詳しく観測できるようになりました。これまでは大腸菌など微生物のモデルをもとにポリメレースがDNA上に動員されると考えられていましたが、ヒトの細胞では、ポリメレースが集まったファクトリーが形成され、そこに沢山の遺伝子が集まってRNAが作られる様子が観察されました。日英の研究チームは、新しい遺伝子制御の基本原理のモデルの樹立に役立ち、特定の遺伝子群でのポリメレースの動きを制御する新しいコンセプトの治療薬開発につながる発見であると考えています。
Youichiro Wada, Yoshihiro Ohta, Meng Xu, Shuichi Tsutsumi, Takashi Minami, Kenji Inoue, Daisuke Komura, Jun'ichi Kitakami, Nobuhiko Oshida, Argyris Papantonis, Akashi Izumi, Mika Kobayashi, Hiroko Meguro, Yasuharu Kanki, Imari Mimura, Kazuki Yamamoto, Chikage Mataki, Takao Hamakubo, Katsuhiko Shirahige, Hiroyuki Aburatani, Hiroshi Kimura, Tatsuhiko Kodama, Peter R. Cook, and Sigeo Ihara
A wave of nascent transcription on activated human genes
PNAS published online before print October 13, 2009